以前要24小时的基因组测序,中国团队只用了7分钟

文章图片
文章图片

文章图片

文章图片

文章图片

文章图片

文章图片

一到年关 , 最不缺的就是各种盘点总结了 。
这不 , 中国机构实现了 7 分钟完成 30X 测序深度人类全基因组测序的成绩 , 时隔 3 个月又被提了起来 。
听不懂没关系 , 我们只需要知道 , 这个成就意味着基因筛查将有可能进入常规体检项 , 遗传病检查也可能像咽拭子检测一样立等可取了 。
比如镰刀型贫血症、先天性心脏病等所有由于基因异常引起的疾病 , 都可以通过基因检测的方式早发现早预防早治疗 , 特别是在生育健康方面意义重大 。
但是目前的基因检查项目大多只针对常见遗传病做筛查 , 一些罕见的遗传病很难被检测到 。 并且检测机构出具报告一般都需要 20 天以上 , 检测项目周期太长 。
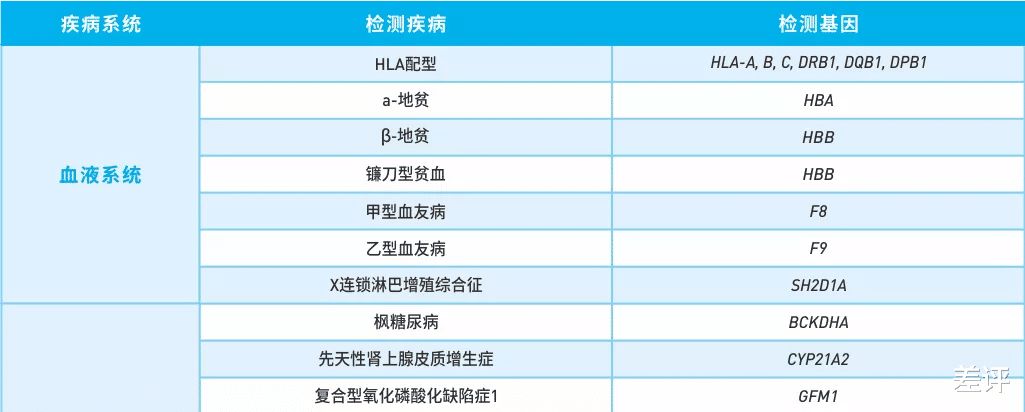
华大医学单基因遗传病检测的部分项目 。 ▼
中国团队把人类全基因组测序所需要的时间 , 直接压缩到了 7 分钟 , 相当于给生物学界开通了一辆和谐号 , 得到生物的全部遗传信息 , 那都是分分钟的事 。
想知道 7 分钟的意义有多大 , 那就先来搞清楚全基因组测序是什么吧 。
基因测序就是把 DNA 信息转换成人类可读取的数字信息过程 , 而全基因组测序 , 就是把生物的所有 DNA 信息全部转化为数字信息 。
读取一整条 DNA 链的碱基排列信息 , 不仅速度慢 , 而且很容易出错 。 在实际操作过程中 , DNA 长链会被切割成许许多多的小片段并同时进行测序 , 这样可以大大减少测序时间 。
虽然小片段序列信息的获取更快更容易 , 但是这也带来了一个新难题 , 如何把这些小片段正确拼接还原成完整序列?
玩过拼图的人都知道 , 判断两块零片是不是相邻位置 , 需要参考它们的图案有没有很好地吻合在一起 。
拼接 DNA 片段也一样 , 两条片段是不是相邻位置 , 要看它们末端的序列能不能完全重叠 。
只要两条序列首尾两端分别存在相同的序列 , 这两段序列就可以合并成一段 。
当然了 , 这是运气好的情况 , 两段相邻片段可以顺利找得出来 。 如果运气不好的话 , 在某一处断点就有可能找不到和它吻合的片段 。
为了保证测序片段能够覆盖整个基因序列 , 常用的手段只有以量取胜 。 把十几倍几十倍的片段往模版里填 , 如果还存在填不上空的情况就该去买彩票了 。
但是片段数量的翻倍直接导致的后果就是拼接工作量的指数增加 , 毕竟拼 1000 块拼图花费的时间可不止是 100 块拼图的十倍 。
这个工作量有多大呢?我们放在具体的测序案例中计算一下 。
以人类全基因组测序为例 , 人类有 23 对染色体共 3.2Gb 碱基对数据 , 一般测序的片段大小会选择在 150-350bp 范围内 , 也就是说 , 对人类基因组测序至少需要处理 10000000 的片段数量 。
而为了提高测序准确率和覆盖度 , 片段的序列数据一般会远超基因组数据 。 比如常用的 30X 测序深度 , 测序得到的总数据达到了基因组数据的 30 倍 , 序列数量大约增加到了 300000000 段 。
粗略估算一下 , 数据读取 300000000 次才能组装好一对小片段 , 第二次组装则需要至少再读取 150000000 次 , 以此类推 。
推荐阅读
- 寻求气候变化的确定性:要多少才足够?
- 跳马冠军桑兰训练瘫痪,33岁冒死产子,母子平安后坦言:想要二胎
- 为什么地球每四年要闰一年?地球相关的冷知识你知道多少
- 癌症会传染吗?人类接触癌细胞不会传染!但要小心这3种病毒 。
- 小伙过年时相亲遭遇尴尬事,如遇此类特征的女子,再漂亮也不能要
- 徒弟杨永财发“长文”爆料垂钓大师李大毛,网友喊话:要懂得尊师重道
- 心灵感应也许是人类认识宇宙真相的重要途径
- 一旦准备好初始状态,通过计算机的量子状态演变将由算法要求的序列确定
- 太阳或将进入“冰河时代”,科学家真的要用核弹引燃木星?
- 男子欠债不还,债主带人上门要钱反被判刑,债主有苦说不出