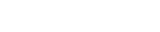近日 , 由中科院海洋所李富花课题组主导完成的科研论文“简单重复序列促进对虾基因组可塑性和适应性进化”在Nature子刊《通讯-生物学》在线发表 。 该研究针对简单重复序列(simple sequence repeat , SSR)在对虾基因组内呈爆发式扩张的现象 , 首次提出SSR在基因组内急速扩张的新机制 , 揭示了SSR在对虾基因组的可塑性和适应性进化过程中的关键作用 。

文章图片
△从简单到复杂
SSR是由1~6个碱基串联重复多次形成 , 在所有已测基因组物种中含量普遍较低(仅约1%左右) , 而且它们通常被认为是没有功能的 , 属于基因组上的“垃圾DNA” 。 然而 , 该课题组前期研究发现凡纳滨对虾基因组SSR含量竟高达23.93% , 引发了研究人员对于SSR在对虾进化中的功能及作用机制的浓厚兴趣 。
文章图片
△对虾基因组SSR扩张与适应性进化
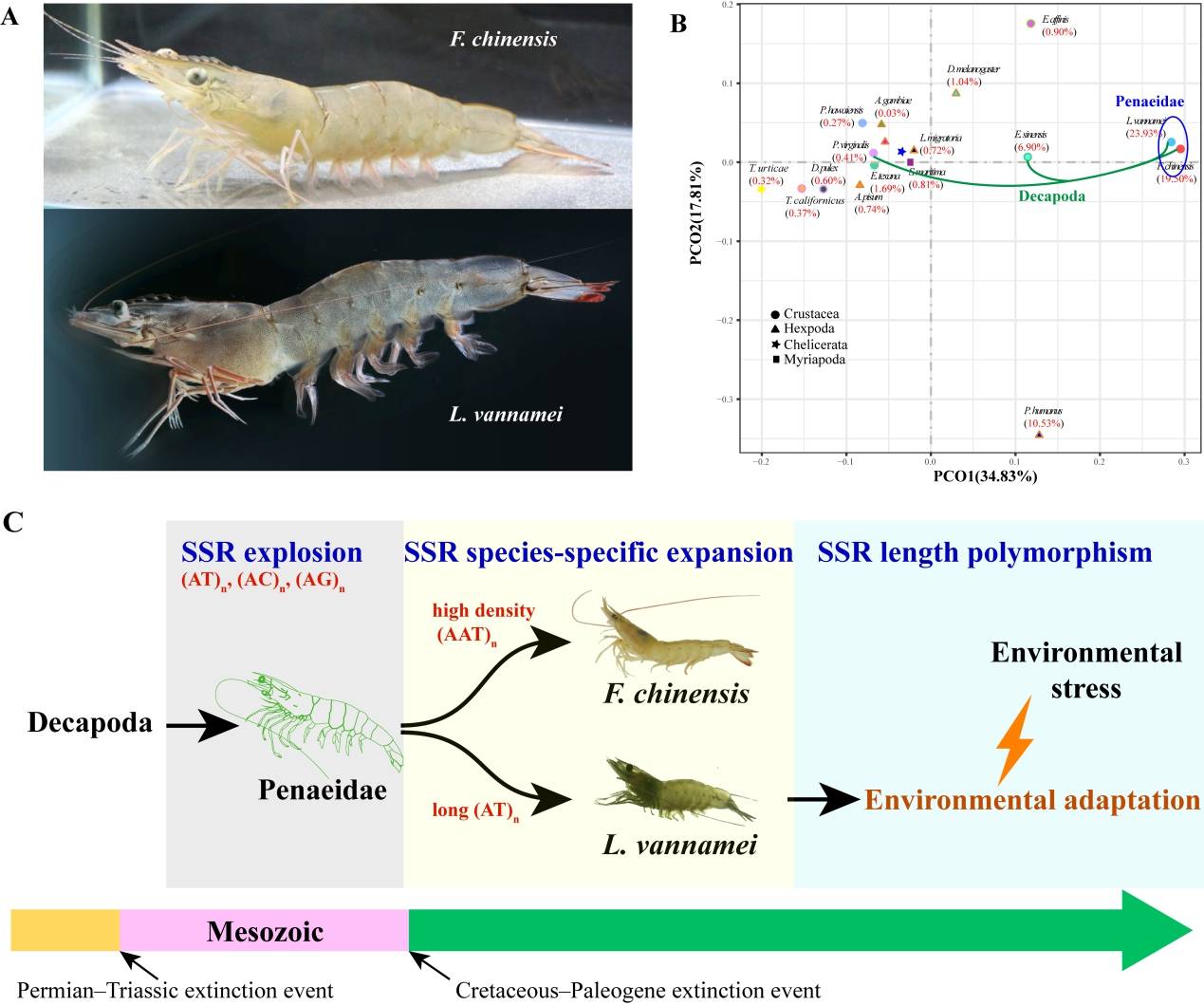
为了探究SSR的分布特征和功能 , 研究人员完成了另一种对虾——中国明对虾——的全基因组测序和组装 , 并基于Hi-C测序 , 构建了凡纳滨对虾和中国明对虾染色体水平的基因组图谱 。 通过比较基因组学分析 , 推断SSR的爆发式扩张发生在对虾的祖先基因组上 , 而SSR的特异性扩张也出现在不同对虾分化之后 。 SSR的扩张事件也与生物大灭绝事件后对虾的快速进化时间一致 , 提示了SSR在对虾适应性进化过程中的关键作用 。
文章图片
△对虾SSR扩张新机制
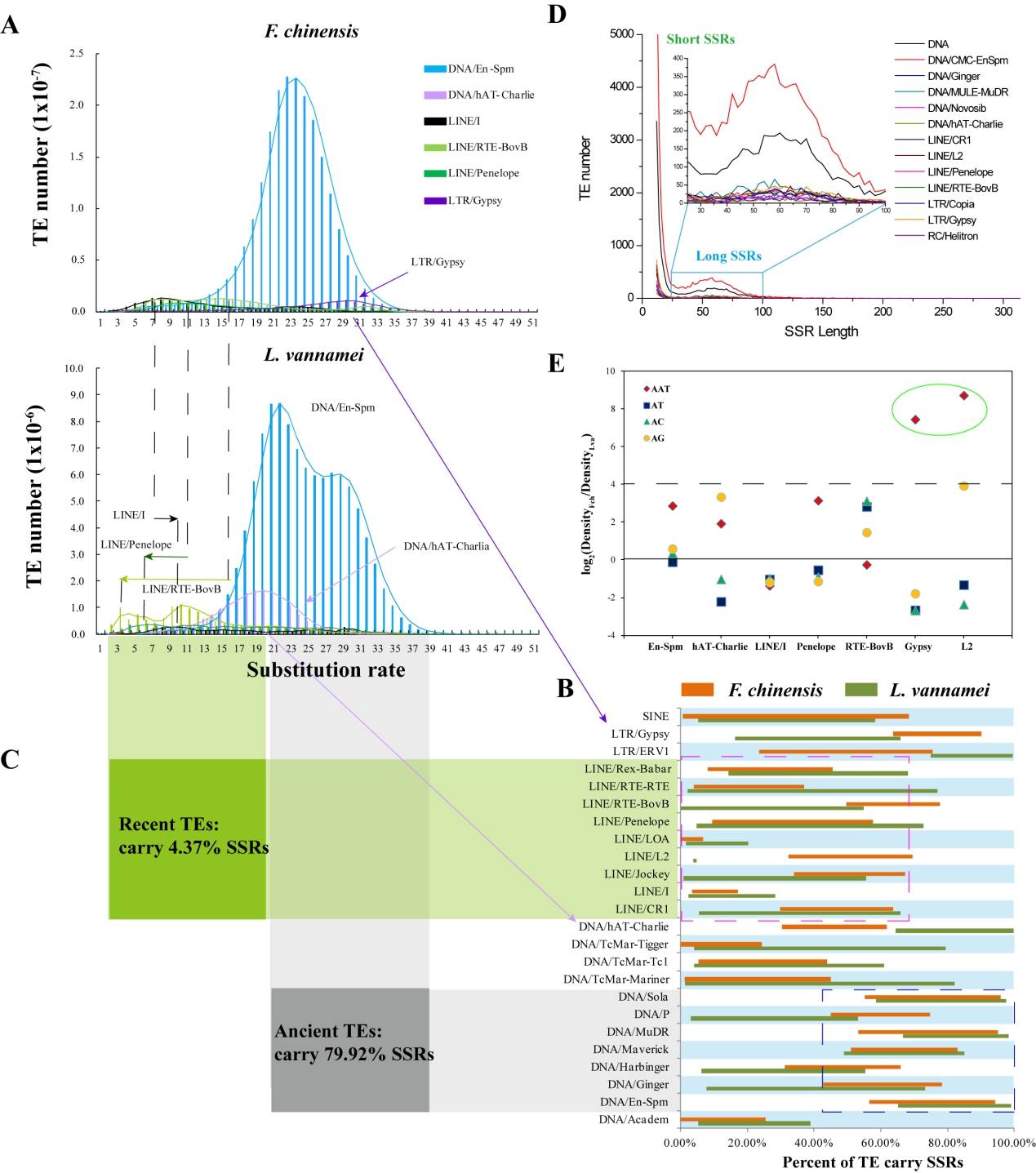
【基因组|新发现!中科院海洋所揭秘对虾进化中的关键因素】本研究首次发现SSR爆发式扩张的新机制 , 与传统认为的复制滑动突变模型不同 , 研究人员发现对虾SSR的爆发式扩张主要与转座元件的携带有关 , 并进一步鉴定出近期活跃的转座元件 , 推断其仍具有携带SSR扩张的潜能 。 此外 , 本研究发现SSR在对虾基因组可塑性和环境适应中具有重要作用 , 高密度的SSR分布引起对虾染色体内大规模基因组重排 。
文章图片
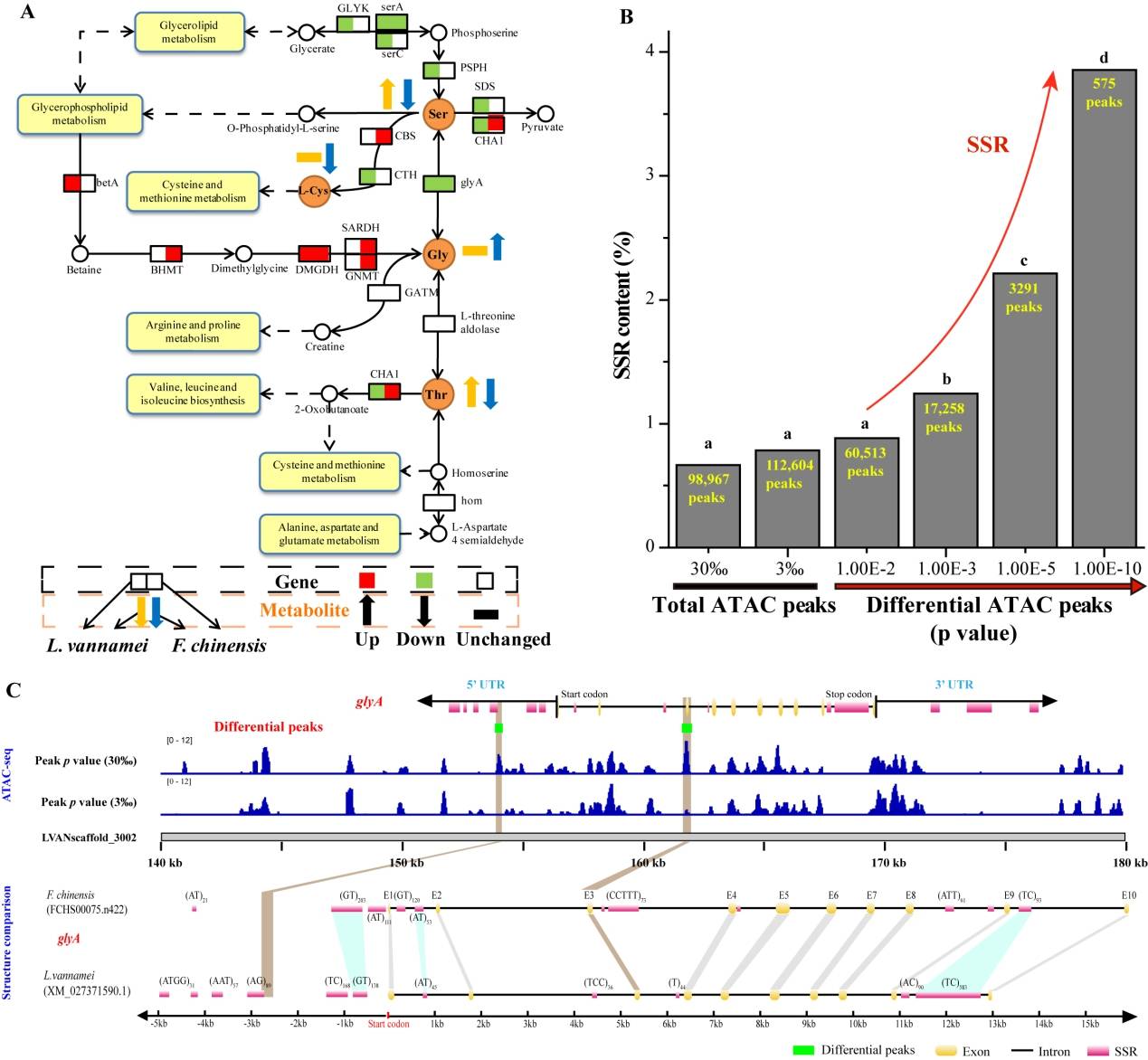
△SSR参与对虾盐度适应相关基因的表达调控
尽管中国明对虾和凡纳滨对虾亲缘关系相近 , 但是它们在盐度适应能力上却存在显著差异 。 研究人员首次发现对虾主要通过调控体内自由氨基酸的含量来调节体内的渗透压 , 而SSR能富集于基因调控区 , 参与渗透压调节关键通路的基因表达调控 。 这些基因表达模式的差异可能是引起两种对虾渗透压调控能力差异的重要原因 。
该成果为阐明生物体内SSR的功能和甲壳动物对环境的适应进化研究提供了新的线索 , 也为对虾基因组育种和分子改良提供了重要的理论基础和数据支撑 。 (总台央视采访人员 王伟)
推荐阅读
- 团队|玉米和水稻基因组引导编辑效率提高3倍
- 编辑效率|玉米和水稻基因组引导编辑效率提高3倍
- 足迹|远古发现丨2021年这些有关“远古”的新发现,哪个曾让你大吃一惊?
- 足迹|2021这些有关“远古”的新发现 哪个让你大吃一惊?
- 化石|远古发现丨2021年这些有关“远古”的新发现,哪个曾让你大吃一惊?
- 材料|中科院成功研制出 4D 打印血管支架
- 青少年|中科院老科学家科普演讲走进三亚中小学校
- 重整|中科院 3D 打印制备车载甲醇重整制氢催化剂研究取得新进展
- 通信技术|中国团队首次将人类全基因组分析缩短至分钟级 此前需要24小时
- 风险|中科院院士王建宇:商业航天很火但风险也比较大