本研究拓展了变构调节在核酸纳米结构中的应用 , 建立了新的调节模型 , 确立了筛选流程 , 丰富了调节机理和完成方式 , 对今后DNA变构元件的研究以及应用具有重要的意义 。
该研究成果由生命学院魏迪明分子设计课题组(MADlab)完成 , 论文题为“基于三螺旋DNA设计纳米变构元件”(Rational design of allosteric nanodevices based on DNA triple helix) , 于10月4日在线发表于《美国化学学会志》(Journal of the American Chemical Society) 。 生命学院博士生张天晴为本文第一作者及共同通讯作者 , 从项目构思、具体实施到最后论文撰写其全程基本独立完成 , 另一共同通讯作者为生命学院魏迪明副教授 , 他在项目立意和写作方面提供了协助 。 该研究得到国家自然科学基金委、清华-北大生命科学联合中心、清华大学结构生物学高精尖中心等基金资助 。
论文链接:
https://pubs.acs.org/doi/10.1021/jacs.1c07824
交叉信息研究院曾坚阳课题组
成功开发多肽和蛋白质相互作用的深度学习模型
近日 , 清华大学交叉信息研究院曾坚阳课题组成功开发了一个基于序列的多尺度预测多肽和蛋白质相互作用的深度学习模型 。 该研究为多肽和蛋白质相互作用的机制提供了一个高效的预测框架 , 可以在为多肽药物预测结合靶点的同时 , 识别多肽序列上的结合位点 。
多肽和蛋白质的相互作用在生物体内起到关键的作用 , 参与多种细胞过程 , 比如信号传导、基因表达调控、细胞增殖和凋亡 。 识别和解析多肽和蛋白质的相互作用及其机制 , 有助于为多肽药物精准定位靶点 , 并为多肽药物的化学修饰提供信息 , 从而加速多肽药物的研发进程 。
目前主流的计算框架 , 分别基于序列的和基于结构来识别蛋白质和多肽配体的相互作用 。 然而 , 这些方法主要集中于识别蛋白质表面与多肽结合结合的残基 , 无法直接提取多肽序列中的结合残基 。 此外 , 基于结构的方法需要用到三维结构信息 , 但通过传统的实验方法测定得到的蛋白质-多肽复合物的结构非常昂贵且耗时 。 在本项研究中 , 作者提出了名为CAMP的深度学习框架 , 用于同时预测多肽-蛋白相互作用(PepPIs)和识别多肽序列上的结合残基 。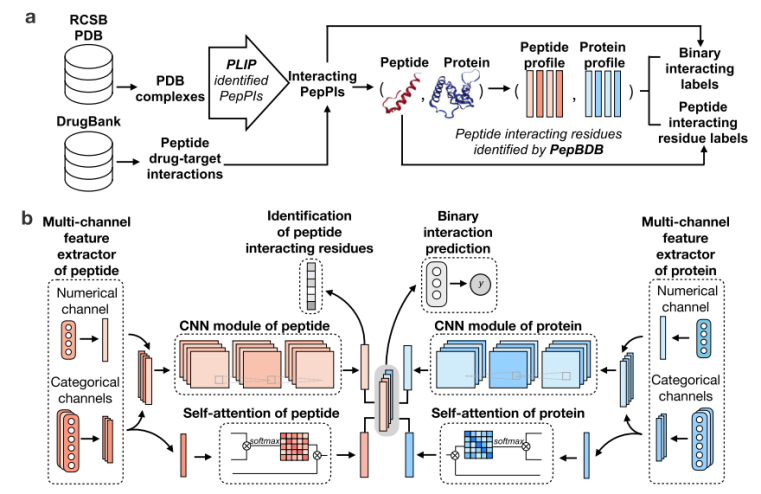
本文图片
CAMP的模型框架图
CAMP将蛋白质和多肽的氨基酸序列、二级结构、理化性质、序列灵活性得分和蛋白质的PSSM矩阵作为模型输入 , 利用卷积神经网络(CNN)模块和自注意力机制(self-attention)来预测给定的肽-蛋白对之间是否存在相互作用 , 同时识别多肽序列上的结合位点 。
推荐阅读
- 载体|可储氢的“纳米巧克力”结构创建
- 油污|纳米涂层的电子烟咪头防水防尘防油污的应用
- 微处理器|碳纳米管“变身”超微型晶体管 宽度仅为人类头发丝的1/25000
- 磁场|利用磁场任意切换纳米激光器的状态
- 电机|1799元,小米米家波轮洗衣机尊享版10kg:纳米银离子除菌率96%
- TSMC|台积电将于明年四季度量产3纳米芯片
- 传播效应|DARPA选择伊庇鲁斯公司开发快速计算电磁传播效应的软件
- 产品|字节入局音乐流媒体,“算法推荐”会带来新“鲶鱼效应”吗?
- 技术|《全球能源基础设施碳排放及锁定效应》报告发布
- 技术|3D打印纳米磁铁揭示磁场中的图案世界